概要
本サンプルはFortran言語によりLAPACKルーチンZPBSVを利用するサンプルプログラムです。
以下の式を解きます。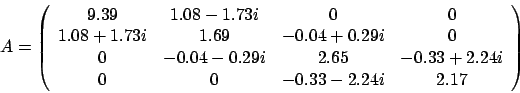
及び
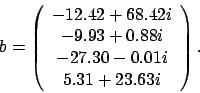
入力データ
(本ルーチンの詳細はZPBSV のマニュアルページを参照)| このデータをダウンロード |
ZPBSV Example Program Data
4 1 :Values of N and KD
( 9.39, 0.00) ( 1.08,-1.73)
( 1.69, 0.00) ( -0.04, 0.29)
( 2.65, 0.00) ( -0.33, 2.24)
( 2.17, 0.00) :End of matrix A
(-12.42,68.42) ( -9.93, 0.88) (-27.30,-0.01) ( 5.31,23.63) :End of vector b
出力結果
(本ルーチンの詳細はZPBSV のマニュアルページを参照)| この出力例をダウンロード |
ZPBSV Example Program Results
Solution
(-1.0000, 8.0000) ( 2.0000,-3.0000) (-4.0000,-5.0000) ( 7.0000, 6.0000)
Cholesky factor U
1 2 3 4
1 ( 3.0643, 0.0000) ( 0.3524,-0.5646)
2 ( 1.1167, 0.0000) (-0.0358, 0.2597)
3 ( 1.6066, 0.0000) (-0.2054, 1.3942)
4 ( 0.4289, 0.0000)
ソースコード
(本ルーチンの詳細はZPBSV のマニュアルページを参照)※本サンプルソースコードのご利用手順は「サンプルのコンパイル及び実行方法」をご参照下さい。
| このソースコードをダウンロード |
Program zpbsv_example
! ZPBSV Example Program Text
! Copyright 2017, Numerical Algorithms Group Ltd. http://www.nag.com
! .. Use Statements ..
Use lapack_example_aux, Only: nagf_file_print_matrix_complex_band_comp
Use lapack_interfaces, Only: zpbsv
Use lapack_precision, Only: dp
! .. Implicit None Statement ..
Implicit None
! .. Parameters ..
Integer, Parameter :: nin = 5, nout = 6
Character (1), Parameter :: uplo = 'U'
! .. Local Scalars ..
Integer :: i, ifail, info, j, kd, ldab, n
! .. Local Arrays ..
Complex (Kind=dp), Allocatable :: ab(:, :), b(:)
Character (1) :: clabs(1), rlabs(1)
! .. Intrinsic Procedures ..
Intrinsic :: max, min
! .. Executable Statements ..
Write (nout, *) 'ZPBSV Example Program Results'
Write (nout, *)
! Skip heading in data file
Read (nin, *)
Read (nin, *) n, kd
ldab = kd + 1
Allocate (ab(ldab,n), b(n))
! Read the upper or lower triangular part of the band matrix A
! from data file
If (uplo=='U') Then
Read (nin, *)((ab(kd+1+i-j,j),j=i,min(n,i+kd)), i=1, n)
Else If (uplo=='L') Then
Read (nin, *)((ab(1+i-j,j),j=max(1,i-kd),i), i=1, n)
End If
! Read b from data file
Read (nin, *) b(1:n)
! Solve the equations Ax = b for x
Call zpbsv(uplo, n, kd, 1, ab, ldab, b, n, info)
If (info==0) Then
! Print solution
Write (nout, *) 'Solution'
Write (nout, 100) b(1:n)
! Print details of factorization
Write (nout, *)
Flush (nout)
! ifail: behaviour on error exit
! =0 for hard exit, =1 for quiet-soft, =-1 for noisy-soft
ifail = 0
If (uplo=='U') Then
Call nagf_file_print_matrix_complex_band_comp(n, n, 0, kd, ab, ldab, &
'Bracketed', 'F7.4', 'Cholesky factor U', 'Integer', rlabs, &
'Integer', clabs, 80, 0, ifail)
Else If (uplo=='L') Then
Call nagf_file_print_matrix_complex_band_comp(n, n, kd, 0, ab, ldab, &
'Bracketed', 'F7.4', 'Cholesky factor L', 'Integer', rlabs, &
'Integer', clabs, 80, 0, ifail)
End If
Else
Write (nout, 110) 'The leading minor of order ', info, &
' is not positive definite'
End If
100 Format ((3X,4(' (',F7.4,',',F7.4,')',:)))
110 Format (1X, A, I3, A)
End Program
